Proteine sind die winzigen Arbeitspferde des Lebens, die in jedem lebenden Organismus eine erstaunliche Vielfalt an Aufgaben erfüllen. Ihre komplexen dreidimensionalen Formen bestimmen ihre Funktion – ein Protein könnte Moleküle durch Zellmembranen befördern, sich gegen Eindringlinge verteidigen oder sogar beschädigte DNA reparieren. Die Entschlüsselung, wie sich diese Aminosäureketten zu diesen komplizierten Strukturen falten, ist seit Jahrzehnten eine zentrale Aufgabe der Biologie.
Doch trotz erheblicher Fortschritte bleiben viele Rätsel rund um die Proteinfaltung ungelöst. 🤯 Genaue Computersimulationen sind für das Verständnis dieses Prozesses von entscheidender Bedeutung, bestehende Modelle haben jedoch Probleme mit der Komplexität. Sie arbeiten auf atomarer Ebene und erfordern eine enorme Rechenleistung, die eine realistische Simulation der Proteinfaltung oft unmöglich macht. Diese Einschränkung wird durch die Tatsache verschärft, dass wir nur die Strukturen von etwa 40 % der menschlichen Proteine kennen – eine große Lücke in unserem biologischen Wissen.
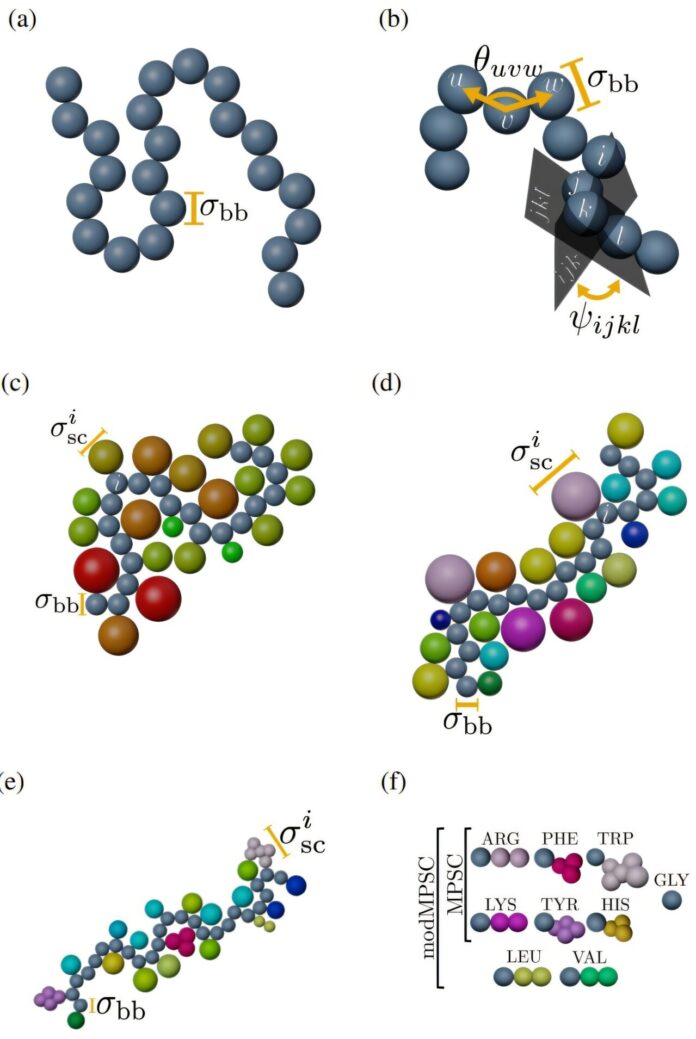
Jetzt haben Forscher der Yale University eine bahnbrechende Lösung entwickelt: bemerkenswert vereinfachte Computermodelle, die wesentliche Proteinmerkmale erfassen, ohne die überwältigenden Details atomarer Simulationen. Diese „grobkörnigen“ Modelle stellen Gruppen von Atomen als einzelne Einheiten dar, wodurch der Rechenaufwand drastisch reduziert wird und gleichzeitig wichtige Strukturinformationen erhalten bleiben.
Das Team testete diese vereinfachten Modelle sorgfältig anhand vorhandener Daten von Tausenden von Proteinen und verglich ihre Vorhersagen mit realen Strukturen und Dichteverteilungen. Sie entdeckten, dass überraschend einfache Darstellungen – eine einzelne Kugel für jede Aminosäure – die zentralen Strukturmerkmale, die zum Verständnis der Proteinfaltung erforderlich sind, effektiv erfassen.
Dieser Durchbruch bedeutet, dass Forscher endlich die Faltung der überwiegenden Mehrheit der Proteine simulieren können, deren Strukturen noch unbekannt sind. 🔓 Dies eröffnet spannende neue Wege in der Arzneimittelentwicklung und Krankheitsforschung. Fehlgefaltete Proteine sind an unzähligen Krankheiten beteiligt, und ein tieferes Verständnis ihrer Faltung könnte den Weg für innovative Therapien ebnen, die diese Missgeschicke an der Wurzel packen.
„Mit diesem grobkörnigen Proteinmodell werden wir in der Lage sein, 60 % der Proteine mit unbekannten Strukturen zu falten“, erklärt Corey O’Hern, Professor für Maschinenbau und Hauptautor der in Physical Review E veröffentlichten Studie. Diese Vereinfachung ermöglicht es Forschern, ein bisher unlösbares Problem anzugehen und bringt uns der Entschlüsselung der Geheimnisse dieser grundlegenden biologischen Maschinen näher.