Les protéines sont les minuscules bêtes de somme de la vie, accomplissant une étonnante variété de tâches au sein de chaque organisme vivant. Leurs formes tridimensionnelles complexes dictent leur fonction : une protéine peut transporter des molécules à travers les membranes cellulaires, se défendre contre les envahisseurs ou même réparer l’ADN endommagé. Décrypter comment ces chaînes d’acides aminés se replient en ces structures complexes est une quête centrale en biologie depuis des décennies.
Pourtant, malgré des progrès significatifs, de nombreux mystères entourant le repliement des protéines restent entiers. 🤯 Des simulations informatiques précises sont cruciales pour comprendre ce processus, mais les modèles existants sont confrontés à la complexité. Ils fonctionnent au niveau atomique, nécessitant une immense puissance de calcul qui rend souvent impossible la simulation réaliste d’un repliement des protéines. Cette limitation est aggravée par le fait que nous ne connaissons que la structure d’environ 40 % des protéines humaines – une vaste lacune dans nos connaissances biologiques.
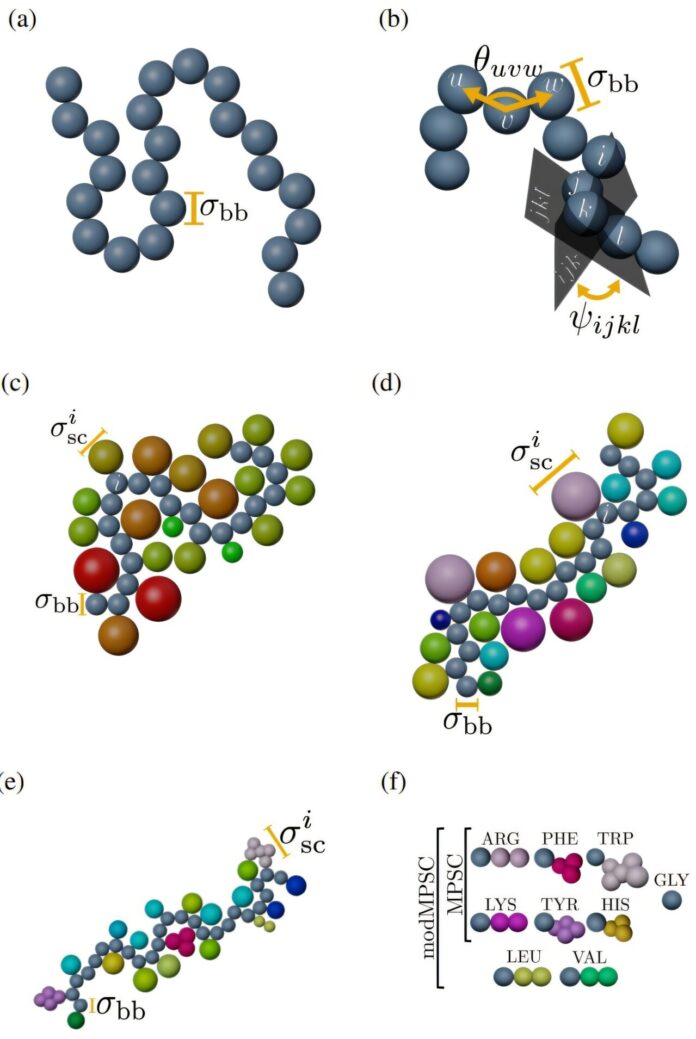
Aujourd’hui, des chercheurs de l’Université de Yale ont développé une solution révolutionnaire : des modèles informatiques remarquablement simplifiés qui capturent les caractéristiques essentielles des protéines sans les détails écrasants des simulations atomiques. Ces modèles « à gros grain » représentent des groupes d’atomes sous forme d’unités uniques, réduisant considérablement les exigences de calcul tout en préservant les informations structurelles clés.
L’équipe a méticuleusement testé ces modèles simplifiés par rapport aux données existantes provenant de milliers de protéines, en comparant leurs prédictions aux structures et distributions de densité du monde réel. Ils ont découvert que des représentations étonnamment simples – une seule sphère pour chaque acide aminé – capturaient efficacement les caractéristiques structurelles essentielles nécessaires à la compréhension du repliement des protéines.
Cette avancée permet aux chercheurs de simuler enfin le repliement de la grande majorité des protéines dont les structures restent inconnues. 🔓 Cela ouvre de nouvelles voies passionnantes dans la découverte de médicaments et la recherche sur les maladies. Les protéines mal repliées sont impliquées dans d’innombrables maladies, et une compréhension plus approfondie de la façon dont elles se replient pourrait ouvrir la voie à des thérapies innovantes ciblant ces incidents à leur cause profonde.
“Grâce à ce modèle protéique à gros grains, nous pourrons replier 60 % des protéines dont les structures sont inconnues”, explique Corey O’Hern, professeur de génie mécanique et auteur principal de l’étude publiée dans Physical Review E. Cette simplification permet aux chercheurs de s’attaquer à un problème auparavant insoluble, nous rapprochant ainsi de la découverte des secrets de ces machines biologiques fondamentales.