As proteínas são os pequenos cavalos de batalha da vida, realizando uma surpreendente variedade de tarefas em cada organismo vivo. Suas complexas formas tridimensionais ditam sua função – uma proteína pode transportar moléculas através das membranas celulares, defender-se contra invasores ou até mesmo reparar DNA danificado. Decifrar como essas cadeias de aminoácidos se dobram nessas estruturas intrincadas tem sido uma questão central na biologia há décadas.
No entanto, apesar do progresso significativo, muitos mistérios que cercam o enovelamento das proteínas permanecem sem solução. 🤯 Simulações computacionais precisas são cruciais para a compreensão desse processo, mas os modelos existentes enfrentam complexidade. Eles operam em nível atômico, exigindo imenso poder computacional que muitas vezes torna impossível simular o dobramento realista de proteínas. Esta limitação é agravada pelo facto de apenas conhecermos as estruturas de cerca de 40% das proteínas humanas – uma grande lacuna no nosso conhecimento biológico.
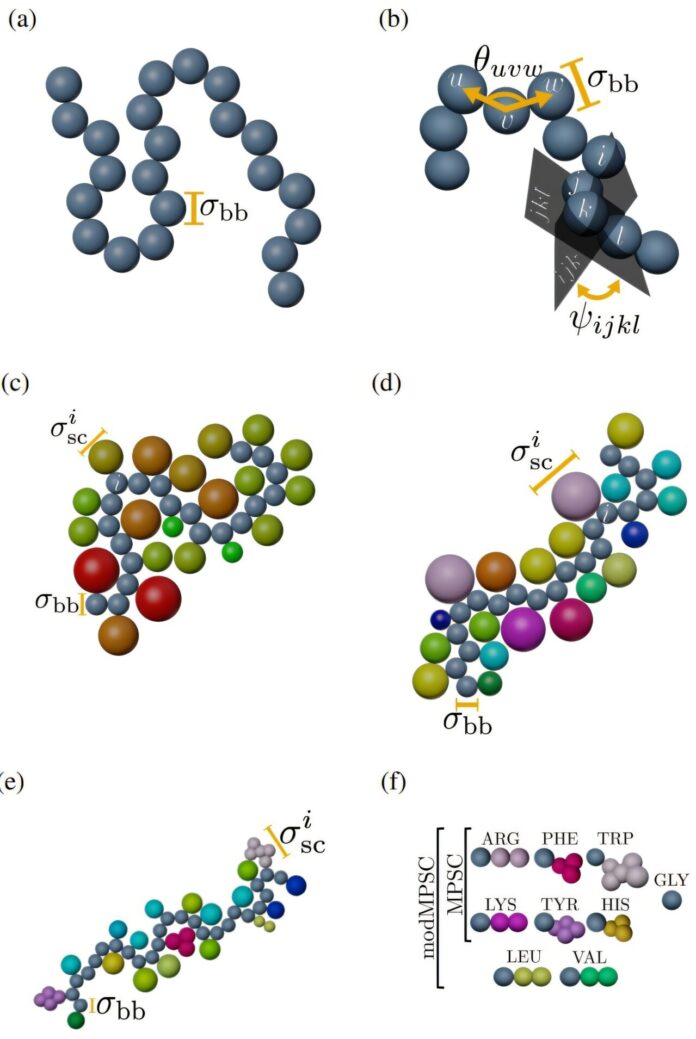
Agora, pesquisadores da Universidade de Yale desenvolveram uma solução inovadora: modelos de computador notavelmente simplificados que capturam características essenciais das proteínas sem os detalhes esmagadores das simulações atômicas. Esses modelos de “granulação grossa” representam grupos de átomos como unidades únicas, reduzindo drasticamente as demandas computacionais e preservando informações estruturais importantes.
A equipe testou meticulosamente esses modelos simplificados em relação aos dados existentes de milhares de proteínas, comparando suas previsões com estruturas do mundo real e distribuições de densidade. Eles descobriram que representações surpreendentemente simples – uma única esfera para cada aminoácido – capturavam efetivamente as principais características estruturais necessárias para a compreensão do enovelamento das proteínas.
Esta descoberta significa que os investigadores podem finalmente simular o enovelamento da grande maioria das proteínas cujas estruturas permanecem desconhecidas. 🔓 Isso abre novos caminhos interessantes na descoberta de medicamentos e na pesquisa de doenças. As proteínas mal dobradas estão implicadas em inúmeras doenças, e uma compreensão mais profunda de como se dobram poderia abrir caminho para terapias inovadoras que visassem estes acidentes na sua causa raiz.
“Com este modelo de proteína de granulação grossa, seremos capazes de dobrar 60% das proteínas com estruturas desconhecidas”, explica Corey O’Hern, professor de engenharia mecânica e principal autor do estudo publicado na Physical Review E. Esta simplificação permite aos investigadores enfrentar um problema anteriormente intratável, aproximando-nos de desvendar os segredos destas máquinas biológicas fundamentais.